R-Pakete
- R ist erweiterbar.
- Für eine große Zahl Anwendungen existieren zusätzliche nützliche Funktionen, die in Paketen organisiert sind.
- Um sie nutzen zu können, muss man das entsprechende Paket zuerst laden.
- Um ein Paket laden zu können muss es installiert sein. Die Installation kann systemweit erfolgen (Administrator) oder in eine persönliche Bibliothek.
R-Pakete installieren
Bevor man Funktionen aus einem Paket aktivieren und benutzen kann muss es ggf. installiert werden.
# Um ein R-Paket zu installieren reicht folgendes
> install.packages('paketname')
# Um ein Installationsverzeichnis explizit anzugeben:
> install.packages('paketname', lib='/home/username/myRlib')
Es gibt immer mindestens zwei Paket-Bibliotheken, "site-wide" (Administrator) und benutzerspezifisch.
CRAN-Mirrors
- Pakete werden standardmäßig über das Internet von einem der CRAN-Spiegelserver installiert.
- CRAN: "Comprehensive R archive network''
- CRAN ist die zentrale Anlaufstelle für veröffentlichte R-Pakete. Um die Serverlast gering zu halten existieren zahlreiche, weltweit verteilte Mirror-Server mit gleichem Inhalt.
- Es gibt noch andere Paketquellen, wie BioConductor oder Github…
Andere Möglichkeiten
- Ein spezialisiertes Paket für den Umgang mit Updates und Softwaremanagement: installr
- Bietet komfortablere Funktionen: updateR(), install.RStudio(), install.pandoc(), uninstall.packages()...
- devtools bietet u.a. Funktionen zur Paketinstallation von Quellen wie Github, Gitlab, Bitbucket, Bioconductor...
- remotes spezialisierter Ersatz für die Installationsfunktionen von devtools
remotes::install_github('repository/paketname') # Z.Bsp. per remotes von GitHub
R-Pakete entfernen
remove.packages('Paketname') entfernt Pakete, egal aus welcher Quelle sie installiert wurden.
Nötigenfalls kann man auch einfach den Paketordner aus der lokalen Paketbibliothek löschen.
R-Pakete installieren
Ein Beispiel:
> install.packages('ggplot2')
Installiere Paket nach '/home/username/R/x86_64-pc-linux-gnu-library/3.4'
(da 'lib' nicht spezifiziert)
– Bitte einen CRAN Spiegel fuer diese Sitzung auswaehlen –
versuche URL 'https://ftp.gwdg.de/pub/misc/cran/src/contrib/ggplot2_2.2.1.tar.gz'
Content type 'application/octet-stream' length 2213308 bytes (2.1 MB)
==================================================
downloaded 2.1 MB
* installing *source* package 'ggplot2' ...
** Paket 'ggplot2' erfolgreich entpackt und MD5 Summen ueberprueft
...
* DONE (ggplot2)
Die heruntergeladenen Quellpakete sind in
'/tmp/RtmpCnOyO7/downloaded_packages'
R-Pakete laden
Ein installiertes Paket kann man folgendermaßen für den Arbeitsbereich aktivieren:
> library(paketname) # keine Anfuehrungszeichen
> library(help=ggplot2) # gibt einen Ueberblick ueber den Paketinhalt
Einige weitere häufig benötigte Befehle
…bzw. Funktionen
- grep()
- gsub()
- apply() & Co.
- length()
- rev()
Weitere Befehle – grep
grep(): Mit der Funktion werden übereinstimmende Muster in einem "character" Vektor gesucht.
Beispiel:
# Im folgenden Bespiel sollen alle "GC" in kurzen Sequenzen gesucht werden
> seq <- c("GTGGGGCATTTACGTGGCT", "AATTAAACATGTAG",
"GCGCAAATAGTCT", "AAAGACAGTGATGACCC")
> grep("GC", seq, perl = TRUE, value = FALSE)
[1] 1 3
Weitere Befehle – grep
Wenn das Argument value = FALSE gesetzt ist oder value nicht spezifiziert ist, gibt grep() die Indices der entsprechenden Elemente zurück.
Wird value = TRUE gesetzt, werden die entsprechenden Elemente ausgegeben.
# Jetzt werden die Elemente ausgegeben
> grep("GC", seq, perl = TRUE, value = TRUE)
[1] "GTGGGGCATTTACGTGGCT" "GCGCAAATAGTCT"
Weitere Befehle – sub
sub() Wie bei grep() werden übereinstimmende Elemente in einem "character" Vektor gesucht. In diesem Fall wird der erste übereinstimmende String ersetzt.
> sub("GC", "GT", seq, perl = TRUE)
[1] "GTGGGGTATTTACGTGGCT" "AATTAAACATGTAG" "GTGCAAATAGTCT"
[4] "AAAGACAGTGATGACCC"
gsub() ersetzt im Gegensatz zu sub() alle übereinstimmenden Elemente.
> gsub("GC", "GT", seq, perl = TRUE)
[1] "GTGGGGTATTTACGTGGTT" "AATTAAACATGTAG" "GTGTAAATAGTCT" "AAAGACAGTGATGACCC"
Weitere Befehle – apply
apply() wendet eine Funkion spalten- oder zeilenweise auf eine Matrix oder ein Array an. Durch dieses vektorwertige Programmieren werden Schleifen umgangen.
Allgemeine Form:
apply(X, MARGIN, FUN, ...)
- mit X Array oder Matrix
- MARGIN einzuhaltende Dimension; in einer Matrix indiziert 1 die Zeilen, 2 die Spalten
- FUN anzuwendende Funktion
Beispiel: Eine Matrix wird definiert. Aus dieser werden die höchsten Werte in allen Spalten ausgelesen:
> X <- matrix(c(2, 10, 8, 5, 18, 7,
9, 4, 17), nrow = 3)
> apply(X, 2, max)
[1] 10 18 17
Weitere Befehle – lapply
lapply() wendet eine Funktion elementweise auf eine Liste, einen dataframe oder einen Vektor an. Eine Liste der Länge des ursprünglichen Objektes wird ausgegeben.
Allgemeine Form:
lapply(X, FUN, ...)
Weitere Befehle – lapply
Z.B.: Wir generieren einen Dataframe mit Expressionswerten für verschiedene Gene (MEDEA; FIS; FIE; PHE; MEI) in zwei Replikaten (rep1 und rep2):
# data.frame mit Gennamen und Expressionswerten
> data <- data.frame(genes = c("MEDEA", "FIS", "FIE", "PHE", "MEI"),
expr_rep1 = c(122, 1028, 10, 458, 77), exprs_rep2 = c(98, 999, 20, 387, 81),
stringsAsFactors = FALSE)
> data
genes expr_rep1 exprs_rep2
1 MEDEA 122 98
2 FIS 1028 999
3 FIE 10 20
4 PHE 458 387
5 MEI 77 81
Weitere Befehle – lapply
# Im Folgenden wollen wir den Logarithmus zur Basis 2 der Expressionsdaten auslesen
> y <- lapply(data[,2:3], log2)
# Als Ergebnis erhalten wir eine Liste
> y
$expr_rep1
[1] 6.930737 10.005625 3.321928 8.839204 6.266787
$exprs_rep2
[1] 6.614710 9.964341 4.321928 8.596190 6.339850
Weitere Befehle – sapply
Ein einfaches Objekt erhalten wir als Rückgabe, wenn wir statt lapply sapply benutzen. Hier erfolgt die Ausgabe in Form einer Matrix:
> z <- sapply(data[, 2:3], log2)
> z
expr_rep1 exprs_rep2
[1,] 6.930737 6.614710
[2,] 10.005625 9.964341
[3,] 3.321928 4.321928
[4,] 8.839204 8.596190
[5,] 6.266787 6.339850
Die apply-Familie
| Function | Input | Output |
|---|---|---|
| aggregate | dataframe | dataframe |
| apply | array | array/list |
| by | dataframe | list |
| lapply | list | list |
| mapply | arrays | array/list |
| sapply | list | array |
Es ist nicht immer leicht, die richtige Funktion auszuwählen.
Weitere Befehle – length
Gibt die Anzahl Elemente in einem Objekt aus.
# e.g. a vector
> a<-2:11
> length(a)
[1] 10
# e.g. a matrix
> a<-matrix(1:12,nrow=3) # e.g. matrix
> a
[,1] [,2] [,3] [,4]
[1,] 1 4 7 10
[2,] 2 5 8 11
[3,] 3 6 9 12
> length(a)
[1] 12
NB.…
# e.g. dataframe
> b<-data.frame(1:2, 2:3)
> b
X1.4 X2.5
1 1 2
2 2 3
> length(b)
[1] 2
Weitere Befehle – rev
Kehrt die Reihenfolge der Elemente eines Objekts um.
# vector
> a<-1:11
> a
[1] 1 2 3 4 5 6 7 8 9 10 11
> rev(a)
[1] 11 10 9 8 7 6 5 4 3 2 1
# matrix
m<-matrix(1:12,nrow=3)
> m
[,1] [,2] [,3] [,4]
[1,] 1 4 7 10
[2,] 2 5 8 11
[3,] 3 6 9 12
> rev(m)
[1] 12 11 10 9 8 7 6 5 4 3 2 1
Und…
# data frame
> rev(data)
exprs_rep2 expr_rep1 genes
1 98 122 MEDEA
2 999 1028 FIS
3 20 10 FIE
4 387 458 PHE
5 81 77 MEI
Noch mehr Funktionen!
- round(), floor(), ceiling(): Zahlen runden.
- subset()
- %in%
- replace()
round() et al.
> round(1.3)
[1] 1
> floor(1.3)
[1] 1
> ceiling(1.3)
[1] 2
subset()
b <- data.frame(x=1:10,y=11:20,z=21:30)
> b
x y z
1 1 11 21
2 2 12 22
...
> subset(b,y>13)
x y z
4 4 14 24
5 5 15 25
6 6 16 26
...
replace()
> a <- 1:10
> a>4
[1] FALSE FALSE FALSE FALSE TRUE TRUE TRUE TRUE TRUE TRUE
> replace(a, a>4, 99)
[1] 1 2 3 4 99 99 99 99 99 99
%in%
> a <- 1:10
> 1%in%a
[1] TRUE
> 11%in%a
[1] FALSE
Bereiche der Statistik
Statistik setzt sich im Wesentlichen aus zwei Bereichen zusammen:- beschreibende Statistik Die beschreibende Statistik hat zum Ziel, empirische Daten durch Grafiken, Tabellen und Kennzahlen übersichtlich zu beschreiben und zu sortieren
- schliessende Statistik Die schliessende Statistik hat zum Ziel, aus Daten einer Stichprobe Eigenschaften einer Grundgesamtheit abzuleiten. Mathematische Methoden werden für Vorhersagen, Generaliserungen und Schätzungen verwendet.
Deskriptive Statistik
Beschreibende Statistik: Zusammenfassen, Ordnen und Beschreiben von Daten.
- grafisch - in den folgenden Folien zusammengefasst
- nummerisch - wird danach behandelt
Diagrammtypen
- Tortendiagramm
- Balkendiagramm
- Histogramm
- Scatterplot
Grafiken
Beispiele für häufig zur Beschreibung von Daten eingesetzte Grafiken sind Boxplots, Pies, Histogramme
R bietet:
- in R implementierte Funktionen für Grafiken, einige werden wir in der Folge kennenlernen
- spezielle Pakete, die das Erstellen von Grafiken ermöglichen, beispielsweise die "library" lattice
(https://cran.r-project.org/web/packages/lattice/lattice.pdf)
Pie-Chart
Ein Pie-Chart dient zur Darstellung von Werten als Stücke eines Kreises in unterschiedlichen Farben.
Als Grundfunktion dient die Funktion pie()
Allgemeine Form:
> pie(x, labels, radius, main, col, clockwise)
- x: Vektor der Daten
- labels: Beschreibung der Teilstücke
- radius: Radius der Teilstücke mit Werten zwischen -1 und 1
- main: Titel des Charts
- col: Farben der Farbpalette
- clockwise: TRUE – im Uhrzeigersinn, FALSE – entgegen
Tortenbeispiel
# Im folgenden Beispiel werden die Bebauung des Landes von
# Oekogemuesebauer Ernst mit verschiedenem Gemuese in % der
# Gesamtflaeche angegeben
# Generieren der Daten
> x <- c(5, 10, 7, 3, 4, 68)
> labels <- c("Zucchini", "Tomaten", "Gurken", "Feldsalat", "Rosenkohl", "Mais")
# Benennen der Datei und Wahl des Grafikformats
> png(file = "Ernsts_Acker.png")
# Generieren des Pies
> pie(x, labels, clockwise = TRUE)
# Speichern der Grafik
> dev.off()
Tortenbeispiel

Tortenbeispiel
Jetzt möchten wir die Grafik noch benannt haben und andere Farben wählen.
# Generieren der Daten
> x <- c(5, 10, 7, 3, 4, 68)
> labels <- c("Zucchini", "Tomaten", "Gurken", "Feldsalat", "Rosenkohl", "Mais")
# Benennen der Datei
> png(file = "Ernsts_Acker_neu.png")
# Generieren des Pies
> pie(x, labels, main = "Ernsts Acker", col = rainbow(length(x)))
# Speichern der Grafik
> dev.off()
Tortenbeispiel

Tortenbeispiel
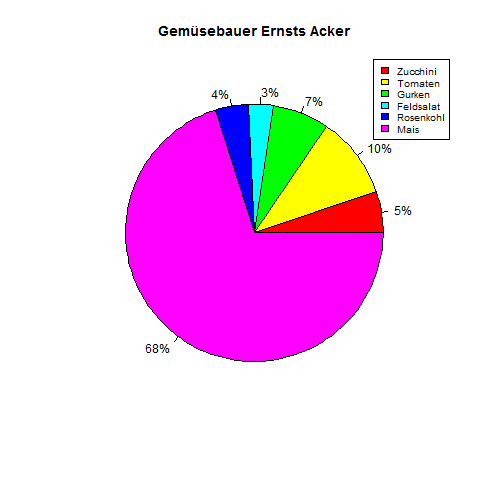
Jetzt sollen die Prozentangaben an den Stücken des Pies stehen und eine Legende in der rechten oberen Ecke eingefügt werden.
> x <- c(5, 10, 7, 3, 4, 68)
> labels <- c("5%", "10%", "7%", "3%", "4%", "68%")
>
> png(file = "Ernsts_Acker_Anteile.png")
>
> pie(x, labels, main = "Gemuesebauer Ernsts Acker",col = rainbow(length(x)))
> legend("topright",
c("Zucchini", "Tomaten", "Gurken", "Feldsalat", "Rosenkohl", "Mais"),
cex = 0.8, fill = rainbow(length(x)))
> # Save the file.
> dev.off()
Tortenbeispiel
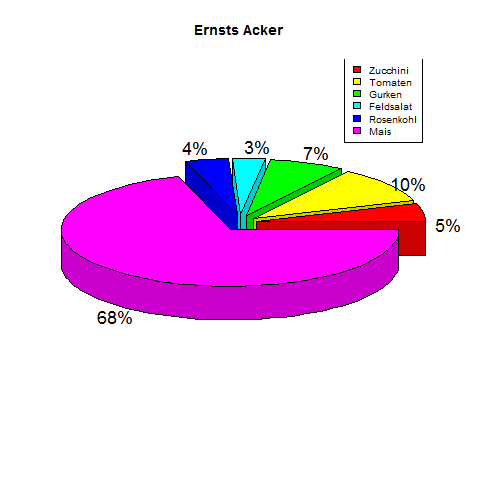
3D-Torten!
Das Ganze geht auch in 3D! Dazu benötigen wir die library "plotrix".
# Laden des Packets plotrix
> library(plotrix)
> # Generieren der Daten
> x <- c(5, 10, 7, 3, 4, 68)
> labels <- c("5%", "10%", "7%", "3%", "4%", "68%")
>
> # Benennen des Charts
> png(file = "Ernsts_Acker_3D.png")
>
> # Plotten.
>
> pie3D(x,labels = labels , explode = 0.1, main = "Ernsts Acker")
> legend("topright",
c("Zucchini", "Tomaten", "Gurken", "Feldsalat", "Rosenkohl", "Mais"),
cex = 0.8, fill = rainbow(length(x)))
> # Save the file.
> dev.off()
Tortenbeispiel
Balkendiagramm
Eine andere Möglichkeit der Darstellung von Daten ist das Balkendiagramm. In R wird es durch die Funktion barplot() generiert.
Allgemeine Form:
barplot(H, xlab, ylab, main, names.arg, col)
- H Vektor der Daten
- xlab Beschriftung der x Achse
- ylab Beschriftung der y Achse
- main Beschriftung des Graphen
- names.arg Vektor der Beschriftungen unter den Balken
- col Farbe(n)
Balkenbeispiel
Wir kommen zurück zu Ernst Acker und stellen die Anbauflächen in seinem Acker prozentual als Balkendiagramm dar.
> # Generieren der Daten
>
> H <- c(5, 10, 7, 3, 4, 68)
> M <- c("Zucchini", "Tomaten", "Gurken", "Feldsalat", "Rosenkohl", "Mais")
>
> # Benennen des Balkendiagramms
> png(file = "Ernsts_Acker_Balkendiagramm.png")
>
> # Generieren des Diagramms
>
> barplot(H,names.arg = M,xlab = "Feldfruechte", ylab = "Anbauflaeche in %",
col = "gray", main = "Ernsts Acker Balkendiagramm", border = "black")
> # Speichern des Diagramms
> dev.off()
Balkenbeispiel

Histogramm
Ein Histogramm ist ein Diagramm, dass die Verteilung einer Variablen darstellt. Die einfachste Art, sich einen Überblick über eine Verteilung von Variablen zu verschaffen, ist sie anzusehen, ohne vorher Berechnungen anzustellen. Um ein Histogramm zu erstellen rufen wir in R die Funktion hist() auf.Histogramm
Allgemeine Form
hist(v,main,xlab,xlim,ylim,breaks,col,border)
mit
- v: Vektor mit nummerischen Werten, die im Histogramm genutzt werden
- main: Beschriftung des Histogramms
- xlab: Beschriftung der x Achse
- ylab: Beschriftung der y Achse
- xlim: beschreibt den Bereich der Werte auf der x Achse
- ylim: beschreibt den Bereich der Werte auf der y Achse
- breaks: legt die Breite der einzelnen Balken fest
- col: legt die Farbe fest
- border: legt die Farbe der Umrandung der Balken fest
Histobeispiel
Um ein Histogramm zu erstellen simulieren wir zunächst ein paar Daten. Hierzu nehmen wir 1000 Werte aus einer Standardnormalverteilung. Das geschieht mit:
> x <- rnorm(1000)
> hist(x)
Histobeispiel
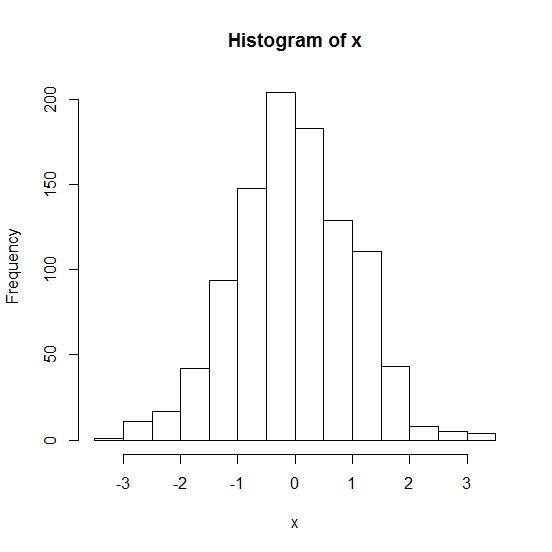
Da wir keine Datei ausgewählt haben sollte die Grafik auf dem Bildschirm ausgegeben werden.
Histobeispiel
Nun fügen wir dem Histogramms einen Titel und Achsenbeschriftungen hinzu. Dazu werden wir noch einen Rahmen um das Histogramm erstellen:
# Im Folgenden erstellen wir aus den Zufallszahlen ein Histogramm mit Beschriftungen
> hist(x, main = "Histogramm Beispiel", xlab = "Zufallszahlen", ylab = "Anzahl")
# Nun wird ein Rahmen um das Histogramm hinzugefuegt
> box()
Histobeispiel
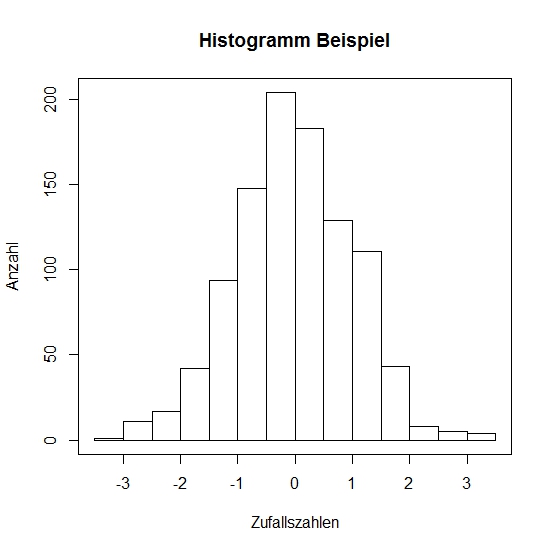
Histobeispiel
Um das Histogramm zu verfeinern, möchten wir nun die Farbe der Balken definieren, das erfolgt mit col. Dazu legen wir noch eine feinere Aufteilung der x-Achse in Intervalle fest. Das geschieht mit breaks. Mit breaks = 50 legen wir fest, dass die x-Achse in 50 Intervalle geteilt ist.
# Hier der Code
> hist(x, main = "Histogramm Beispiel", xlab = "Zufallszahlen",
ylab = "Anzahl", col = "deepskyblue", breaks = 50)
> box()
Histobeispiel

Liniendiagramm
Mit R lassen sich Daten auch als einfache Liniendiagramme darstellen. Das geschieht durch die Funktion plot(). In der allgemeinen Form plot(v,type,col,xlab,ylab) kann man über type = "p", type = "l" oder type = "o" zwischen der Darstellung von nur Punkten ("p"), nur Linien ("l") oder beidem ("o") wählen. Beispiel:
> linie <- c(27:7)
> plot(linie, type = "o", col = "deepskyblue", xlab = "x", ylab = "y")
Scatterplot
Auch ein Scatterplot wird mit der Funktion plot() erstellt, in der allgemeinen Form plot(x, y, main, xlab, ylab, xlim, ylim, axes). Als Beispiel wählen wir den Datensatz "mtcars'', der in der R Umgebung vorhanden ist.
>?mtcars
mtcars {datasets} R Documentation
Motor Trend Car Road Tests
Description
...
Format
A data frame with 32 observations on 11 variables.
[, 1] mpg Miles/(US) gallon
[, 2] cyl Number of cylinders
[, 3] disp Displacement (cu.in.)
[, 4] hp Gross horsepower
[, 5] drat Rear axle ratio
[, 6] wt Weight (1000 lbs)
[, 7] qsec 1/4 mile time
[, 8] vs V/S
[, 9] am Transmission (0 = automatic, 1 = manual)
[,10] gear Number of forward gears
[,11] carb Number of carburetors
...
Scatterplot mtcars
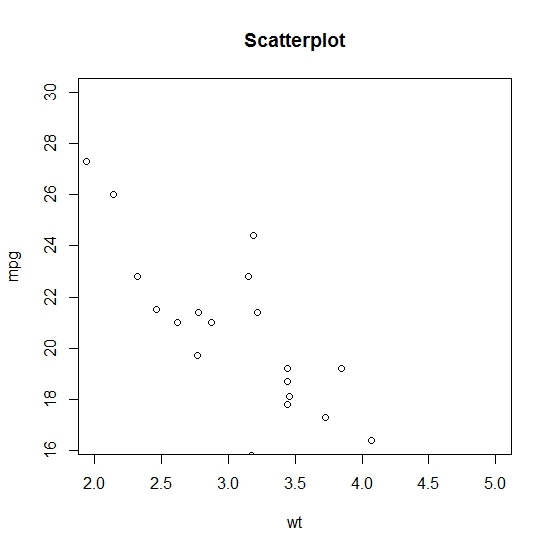
Hier wollen wir nun "mpg" (Verbrauch) und "wt" (Gewicht) vergleichen.
# Erstellen des Dataframes
> input <- mtcars[, c(1,6)]
> head(input)
mpg wt
Mazda RX4 21.0 2.620
Mazda RX4 Wag 21.0 2.875
Datsun 710 22.8 2.320
Hornet 4 Drive 21.4 3.215
Hornet Sportabout 18.7 3.440
Valiant 18.1 3.460
# Bennenen des Plots
> png(file = "scatterplot_mtcars.png")
Scatterplot mtcars
# Plotten von mpg und wt fuer alle Autos mit einem Gewicht zwischen
# 2 und 5 und einer mpg zwischen 16.4 und 30
> plot(x = input$wt, y = input$mpg, main = "Scatterplot",
xlab = "wt", ylab = "mpg", xlim = c(2, 5), ylim = c(16.4, 30))
Scatterplot mtcars
Weitere Methoden der deskriptiven Statistik
Nummerische Zusammenfassung von Daten- Zentrale Tendenzen: Mittelwert, Median, Modus
- Quantile
- Streuungsmaß
Zentrale Tendenz
Mittelwert: Das arithmentrische Mittel wird auch Mittelwert oder Durchschnitt benannt. Es berechnet sich als die Summe aller Einzelwerte geteilt durch die Anzahl.Zentrale Tendenz
- Das arithmetrische Mittel gewichtet alle Zahlen gleich, daher ist es für Ausreißer anfällig.
- Das arithmetrische Mittel ist gut zur Beschreibung von symmetrischen Verteilungen.
- Eine Variante ist der getrimmte Mittelwert. Hier werden extreme Werte, z.B. die größten und kleinsten 10\% für die Berechnung nicht berücksichtigt.
- In R lässt es sich einfach durch mean() berechnen.
Arithmetisches Mittel
# Zunaechst erstellen wir einen Vektor
> x <- c(10, 100, 234, 65, 77, 34, 88, 98, 504, 1)
# Mit mean() berechnen wir den Mittelwert
> mean(x)
[1] 121.1
# Mit der Option trim = 0.1 wird der groesste und der
# kleinste Wert nicht beruecksichtigt
> mean(x, trim = 0.1)
[1] 88.25
Arithmetisches Mittel
Zur Kontrolle:
# Durch sort() wird der Vektor zunaechst sortiert
> y <- sort(x)
# Die groesste und kleinste Zahl wird nicht beruecksichtigt
> mean(y[2:9])
[1] 88.25
Arithmetisches Mittel
Für fehlende Werte kann die Option na.rm = TRUE gesetzt werden
# Zunaechst erstellen wir einen neuen Vektor
> x <- c(10, 100, 234, NA, 65, 77, 34, 88, 98, 504, 1, NA)
# Wegen der fehlenden Zahlen ergibt
> mean(x)
[1] NA
# Jetzt werden die fehlenden Werte nicht beruecksichtigt
> mean(x, na.rm = TRUE)
[1] 121.1
Median (Zentralwert)
Um den Median zu ermitteln, müssen zunächst die Daten der Größe nach geordnet werden. Der Median ist der Wert, der…- …genau in der Mitte liegt, oder…
- …präzise formuliert die Bedinung erfüllt, dass mindestens die Hälfte der Meßwerte kleiner oder gleich bzw. größer oder gleich diesem Wert sind.
Median
In R kann der Median mit der Funktion median() ermittelt werden
# Zunaechst erstellen wir wieder den Vektor
> x <- c(10, 100, 234, 65, 77, 34, 88, 98, 504, 1)
> median(x)
[1] 82.5
Modus
Der Modus ist die Ausprägung oder der Wert, der in einer Verteilung am häufigsten vorkommt. Für den Modus gibt es keine Funktion in R.Können Sie am Ende des Kurses eine schreiben…?
Beschreibung eines Datensatzes
Um zu verstehen, wie weit die Daten streuen (Varianz) schauen wir zunächst, wie unterschiedlich die einzelnen Datenpunkte vom Mittelwert sind.Varianz
Im folgenden Beispiel haben 8 Personen Wichtelgeschenke für Ihre Kollegen gekauft. Die Vereinbarung war, dass die Geschenke 5-10 Euro an Wert haben sollten. Folgender Vektor enthält die von den einzelnen Personen ausgegebenen Beträge in Euro:
> wert <- c(10, 8, 5, 9, 7, 6, 3, 11)
Zunächst schauen wir uns den niedrigsten und höchsten Betrag an:
> range(wert)
[1] 3 11
Es wurden zwischen 3 und 11 Euro ausgegeben. Diese Werte sind stark von möglichen Extremen (Outliern) beeinflusst.
Varianz
Wieviel wurde im Mittel ausgegeben?
# Der Mittelwert der Ausgaben wird mit mean() ermittelt.
> mean(wert)
[1] 7.375
Nun ist interessant, wie weit die einzelnen Werte bzw. Beträge vom Mittelwert jeweils abweichen:
> unterschiede <- wert - mean(wert)
# Abweichungen vom Mittelwert koennen positiv oder
# negativ sein. Daher wird das Quadrat der summierten Werte gebildet.
> unterschiede
[1] 2.625 0.625 -2.375 1.625 -0.375 -1.375 -4.375 3.625
Varianz
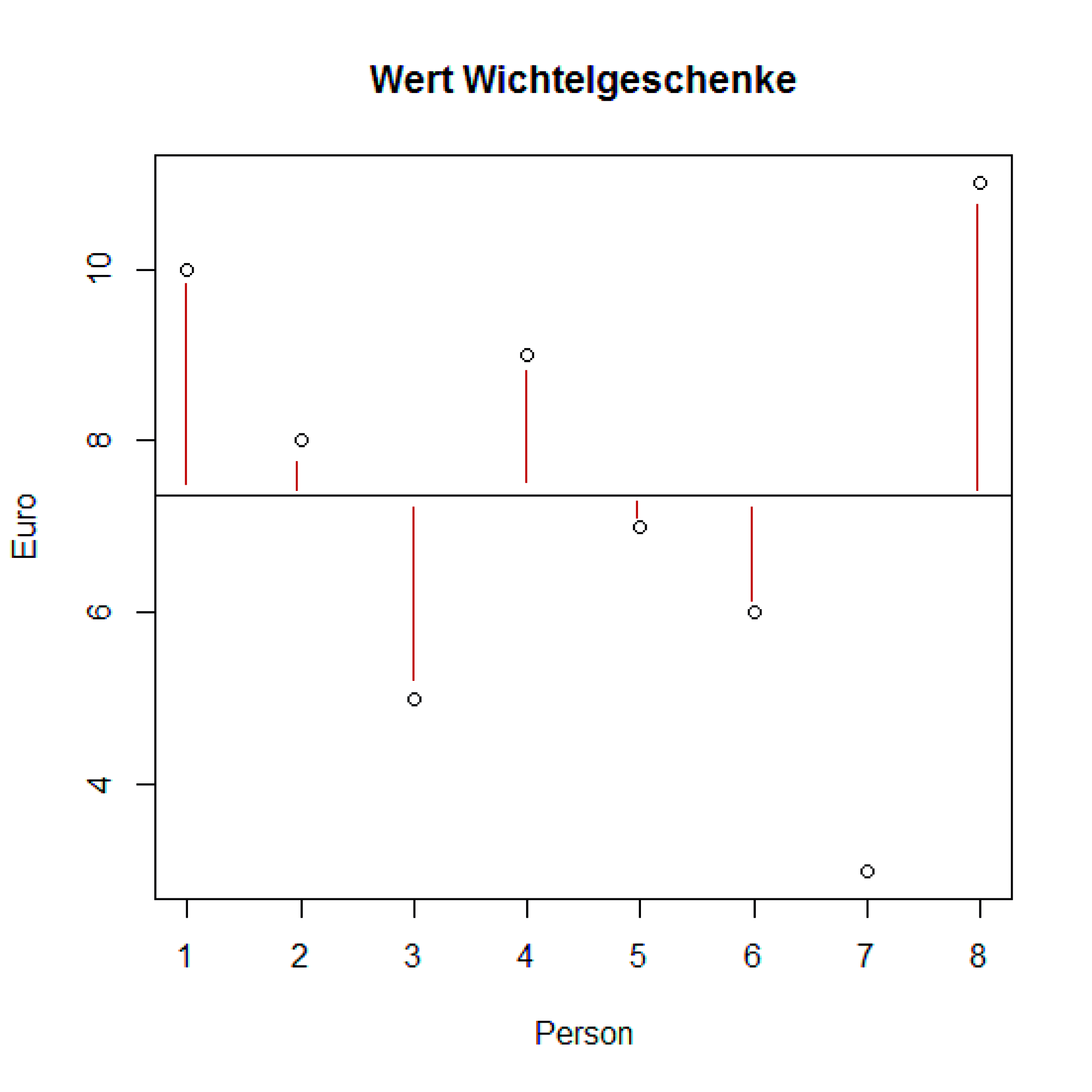
Varianz
Die Varianz ergibt sich nun als Funktion der Summe der Unterschiede im Quadrat durch die Anzahl der Freiheitsgrade.
# Die Funktion wird aufgestellt
> variance <- function(x)sum(((x-mean(x))^2))/(length(x)-1)
# Die Funktion wird ausgefuehrt
> variance(wert)
[1] 7.125
> var(wert)
[1] 7.125
Standardabweichung
Die Varianz hat den Nachteil, dass die Einheiten im Quadrat sind, in diesem Fall also Euro$^2$. Um die Standardabweichung (SD, s) zu erhalten, ziehen wir nun die Quadratwurzel aus der Varianz: $$ s = \sqrt{\frac{1}{n-1}\sum\limits_{j=1}^{n}(x_j-\bar{x})^2} $$In R berechnet sich das sehr einfach mit der Formel sd().
# Jetzt berechnen wir die Standardabweichung
# vom Wert der Wichtelgeschenke:
> sd(wert)
[1] 2.66927
# Nochmals zur Kontrolle die Quadratwurzel der Varianz
> sqrt(var(wert))
[1] 2.66927
Box und Whisker Plot
> boxplot(wert, ylab = "Euro",
main = "Wichtelpreise")
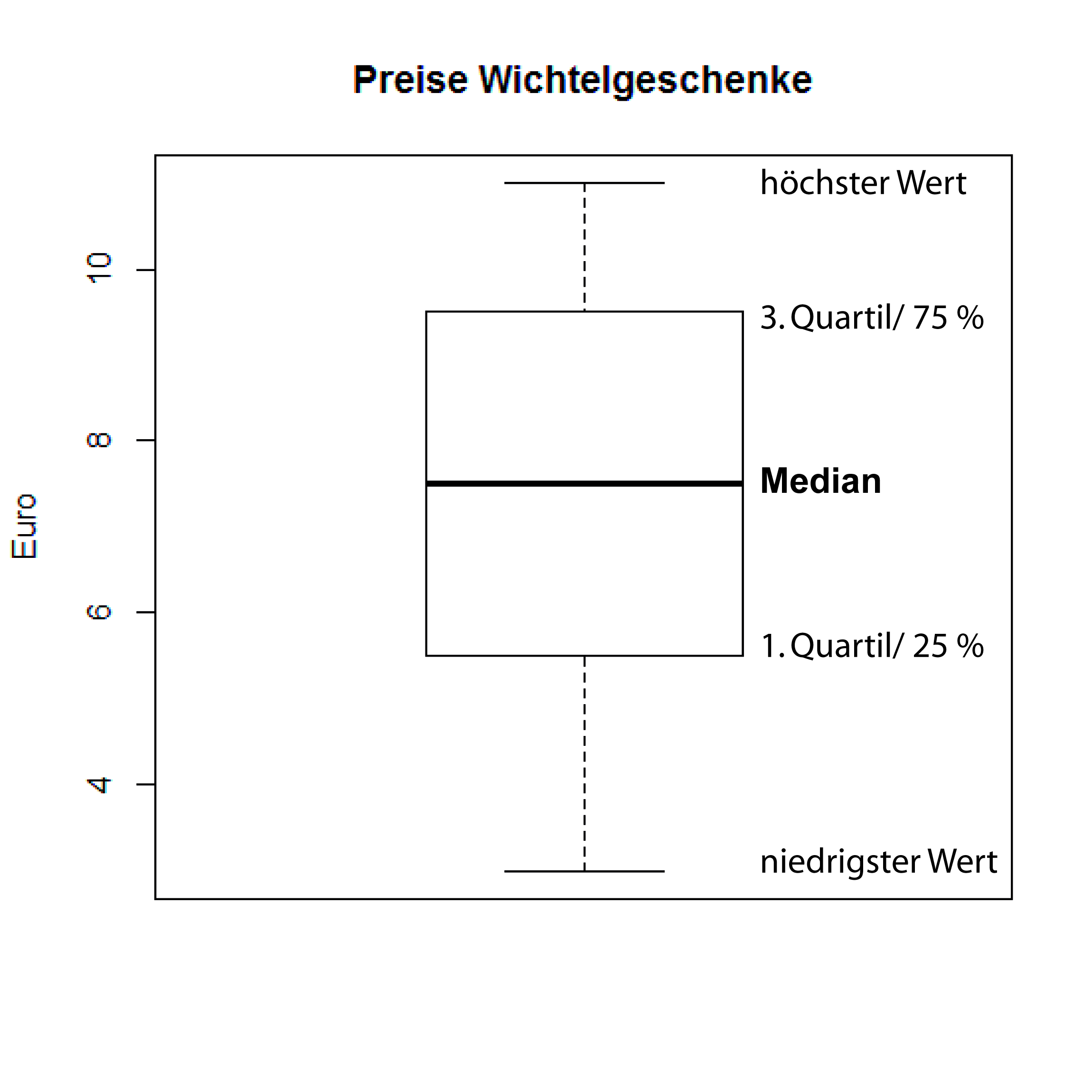
Normalverteilung und Studentische t-Verteilung
Nach dem "Zentralen Grenzwertsatz" (Wahrscheinlichkeitstheorie) sind die Mittelwerte einer großen Zahl unabhängiger Proben ± normalverteilt.
Normalverteilung simuliert
Hier eine Simulation: Wir ziehen 100000mal zufällig fünf Zahlen zwischen 1 und 10 und berechnen den Mittelwert:
# Plot der Mittelwerte von Zufallszahlen
> means <- numeric(100000)
> for (i in 1:100000){
means[i]<-mean(runif(5)*10)
}
> hist(means, ylim = c(0, 16000))
# Mittelwert und Standardabweichung der Probe
# der 100000 Mittelwerte
> mean(means)
[1] 5.0026
> sd(means)
[1] 1.291312
Normalverteilung simuliert
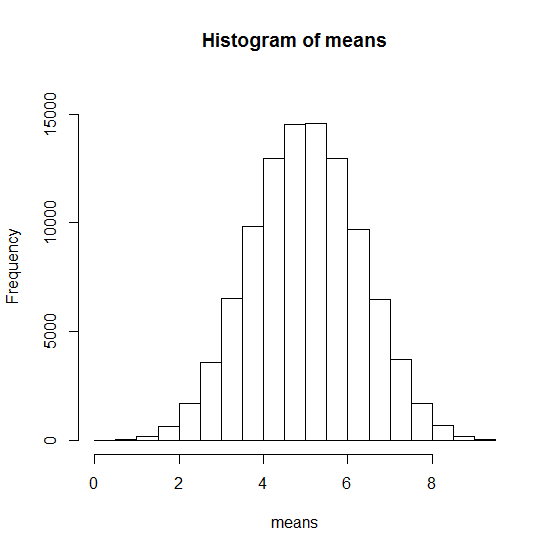
Normalverteilung
Diese Werte werden nun in die Dichtefunktion der Normalverteilung eingesetzt und in den Plot eingebracht:
dnorm(x, mean, sd) # allgemeine Form
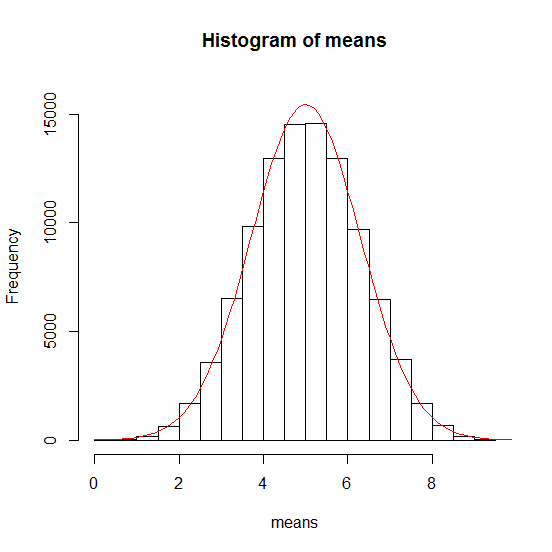
- x: Vektor von Werten
- mean: Mittelwert der Daten der Probe
- sd: Standardabweichung der Mittelwerte
Normalverteilung
# Wir generieren eine Sequenz von Werten zwischen 0 und 10,
# die sich jeweils um 0.1 erhoehen fuer die x-Achse
> xwerte <- seq(0, 10, 0.1)
# Wir geben den Mittelwert und die Standardabweichung fuer
# die Dichteverteilung auf der y-Achse vor
> ywerte <- dnorm(xwerte, mean = 5.0026, sd = 1.291312) * 50000
> lines(xwerte, ywerte, col = "red", type="l")
Normalverteilung versus Student'sche t-Verteilung
Wenn die Stichprobe zu klein ist (n < 30) benötigen wir statt der Normalverteilung die Student'sche t-Verteilung
# Vergleich Normalverteilung und Student'schen
# t-Verteilung fuer eine Probe mit 5 Freiheitsgraden
# (1) Plot der Normalverteilung in blau
> xwerte <- seq(-4, 4, 0.1)
> ywerte <- dnorm(xwerte)
> plot(xwerte, ywerte, type = "l", lty = 1,
col = "deepskyblue", xlab = "Abweichung",
ylab = "Dichteverteilung Wahrscheinlichkeit")
# Erstellen einer t-Verteilung fuer 5 Freiheitsgrade
# (2) gestrichelt plotten in rot
> ywerte <- dt(xwerte, df=5)
> lines(xwerte, ywertet, type = "l", lty = 2, col = "red")
Normalverteilung versus Student'sche t-Verteilung
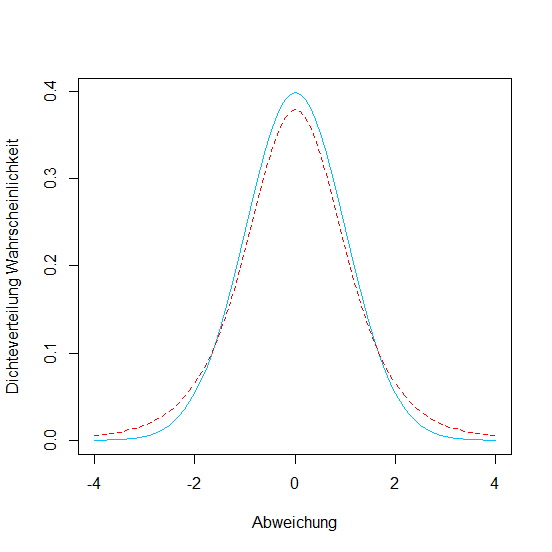
Schließende Statistik
- dient der Vorhersage von Daten und der Beschreibung von Wahrscheinlichkeiten
- aus Stichproben wird auf das Verhalten einer Grundgesamtheit geschlossen
- beinhaltet Hypothesentests und Regressionsmodelle
Voraussagen anhand der Normalverteilung
68% aller Daten fallen in den Bereich des Mittelwertes ± einer Standardabweichung.
# Wir nutzen pnorm() um zu ermitteln wieviel % der Daten
# kleiner sind als der Mittelwert -1 Standardabweichung
> pnorm(-1)
[1] 0.1586553
> 1-pnorm(1)
[1] 0.1586553
Voraussagen anhand der Normalverteilung
Wir möchten nun wissen, innerhalb ± welcher Standardabweichung bei Normalverteilung 95% aller Daten fallen. Dies definiert unser Konfidenzintervall. Dazu nutzen wir qnorm():
> qnorm(c(0.025, 0.975))
[1] -1.959964 1.959964
Voraussagen anhand der Normalverteilung
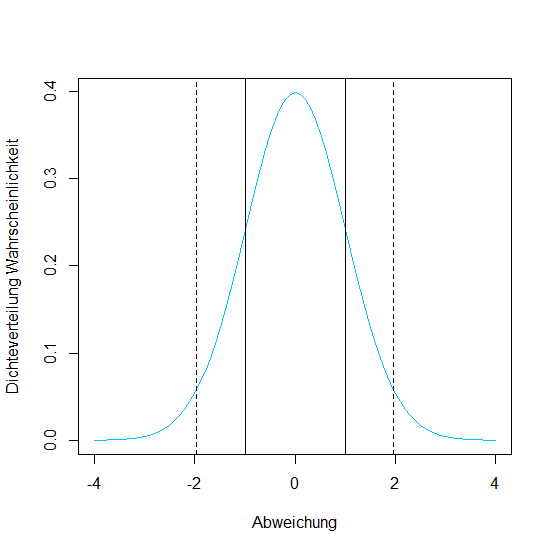
95% aller Daten fallen in den Bereich des Mittelwertes ±1.96 Standardabweichungen bei Normalverteilung!
Voraussagen anhand der Normalverteilung
Wie kann man anhand dessen die Wahrscheinlichkeit voraussagen, dass ein Wert…
- kleiner ist als ein vorgegebener Wert
- größer ist
- oder genau zwischen zwei Werten liegt?
Voraussagen anhand der Normalverteilung
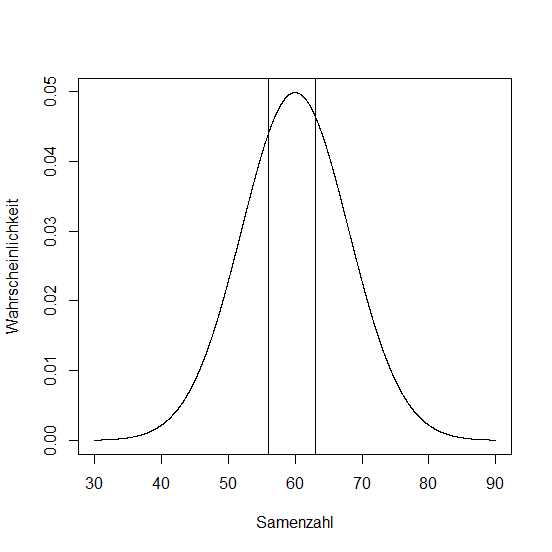
Wir haben bei 600 Schoten von Boechera divaricarpa die Anzahl reifer Samen gezählt. Der Mittelwert war 60, die Standardabweichung 8.
# Ein Plot fuer die entsprechende Normalverteilung
seeds <- seq(30, 90, 0.1)
plot(seeds, dnorm(seeds, 60, 8), type = "l",
xlab = "Samenzahl", ylab = "Wahrscheinlichkeit")
Z Score
- xi: Datenwert
- x̄: Mittelwert
- s: Standardabweichung
> z <- (45-60)/8
> z
[1] -1.875
Z Score
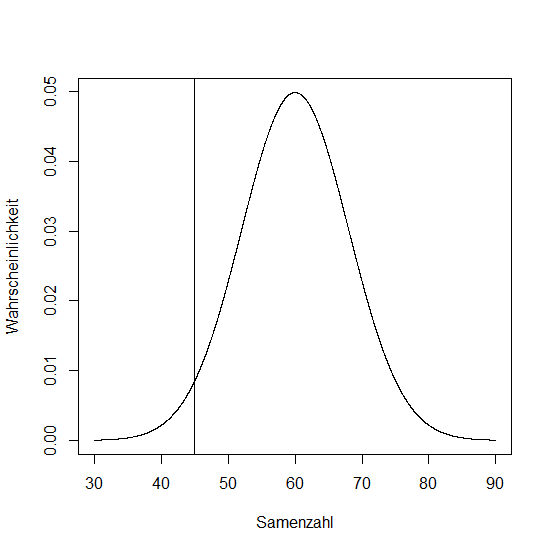
Z Score
Nun bleibt herauszufinden, wie groß die Wahrscheinlichkeit ist, dass die Normalverteilung einen Wert von -1.875 oder kleiner annimmt. Dazu nutzen wir wieder pnorm().
> pnorm(-1.875)
[1] 0.03039636
Damit erhalten wir eine Wahrscheinlichkeit von etwas über 3%.
Z Score
Wie groß ist die Wahrscheinlichkeit, dass eine Schote zwischen 56 und 63 Samen enthält?
> z1 <- (56-60)/8
> z2 <- (63-60)/8
> z1
[1] -0.5
> z2
[1] 0.375
Nun ziehen wir die kleinere von der größeren Wahrscheinlichkeit ab:
> pnorm(0.375)-pnorm(-0.5)
[1] 0.3376322
Die Wahrscheinlichkeit beträgt fast 34%!
Z Score