GIS mit ggplot2
GIS mit ggplot2
Die Ausgangssituation sei ein File mit Sammelpunkten und Angaben zu Datenquelle und per Sequenz bestimmtem Genotyp.
> read.csv('https://lehre.cos.uni-heidelberg.de/presentations/open/R/downloads/
cbpoints.csv') -> pts
> head(pts, 3)
X SequenceType Lat Long DataSource
1 1 1 51.2446 9.2932 PCR
2 2 3A 40.9154 40.9154 PCR
3 3 1 55.6875 9.8683 PCR
Das Ziel ist die Visualisierung dieser Daten auf einer Europa-Karte.
GIS mit ggplot2
Folgende R-Pakete werden gebraucht:
# install.packages(c('sf','ggplot2','rnaturalearth','rnaturalearthhires','rnaturalearthdata'))
library('sf')
library('ggplot2')
library('rnaturalearth')
library('rnaturalearthhires')
library('rnaturalearthdata')
GIS mit ggplot2
Wir brauchen ein SF-Object mit Geo-Daten:
ptsf <- st_as_sf(pts, coords=c('Long', 'Lat'))
Fehler in st_as_sf.data.frame(pts, coords = c("Long", "Lat")) :
missing values in coordinates not allowed
# Ups
...
105 105 1 49.69232 22.4858833 PCR
106 NA NA NA PCR
107 107 1 39.80000 21.1166667 PCR
...
#Aha...
> pts <- pts[!is.na(pts$Lat),]
> ptsf <- st_as_sf(pts, coords=c('Long', 'Lat'))
> sf_use_s2(FALSE) # S2 features abschalten
> st_crs(ptsf) = 4326 # Projektion setzen
GIS mit ggplot2
> ptsf
Simple feature collection with 199 features and 3 fields
Geometry type: POINT
Dimension: XY
Bounding box: xmin: -3.220519 ymin: 36.23028 xmax: 82.53025 ymax: 62.92458
Geodetic CRS: WGS 84
First 10 features:
X SequenceType DataSource geometry
1 1 1 PCR POINT (9.2932 51.2446)
2 2 3A PCR POINT (40.9154 40.9154)
3 3 1 PCR POINT (9.8683 55.6875)
4 4 1 PCR POINT (14.65 58.3333)
5 5 3A PCR POINT (28.9889 41.1836)
...
Punkte sind vorbereitet.
GIS mit ggplot2
Dann brauchen wir noch eine Karte.
eu <- c("Albania","Austria","Belgium","Bulgaria","Belarus","Bosnia and Herzegovina","Croatia","Cyprus",
"Czech Republic","Denmark","Estonia","Finland","France",
"Germany","Greece","Hungary","Ireland","Italy","Latvia",
"Lithuania","Luxembourg","Macedonia","Malta","Moldova","Montenegro","Netherlands","Poland",
"Portugal","Romania","Russia","Slovakia","Republic of Serbia","Slovenia","Spain",
"Sweden","Switzerland","United Kingdom","Ukraine")
euro <- ne_states(country=eu, returnclass = "sf")
w <- ggplot() + geom_sf(data=euro_cropped, col="darkgrey", show.legend=F)
w
GIS mit ggplot2
Nicht schön, als Europakarte, besser zuschneiden.
euro <- ne_countries(scale = "medium", returnclass = "sf")
euro_cropped <- st_crop(euro, xmin = -20, xmax = 45, ymin = 30, ymax = 73)
ptsf_cropped <- st_crop(ptsf, xmin = -20, xmax = 45, ymin = 30, ymax = 73)
w <- ggplot() + geom_sf(data=euro_cropped, col="darkgrey", show.legend=F)
w
GIS mit ggplot2
Besser.
GIS mit ggplot2
Fehlen noch die Punkte.
w <- w + geom_sf(data=ptsf_cropped,
aes(shape=ptsf_cropped$DataSource,col=ptsf_cropped$SequenceType),
show.legend=T)
w
GIS mit ggplot2
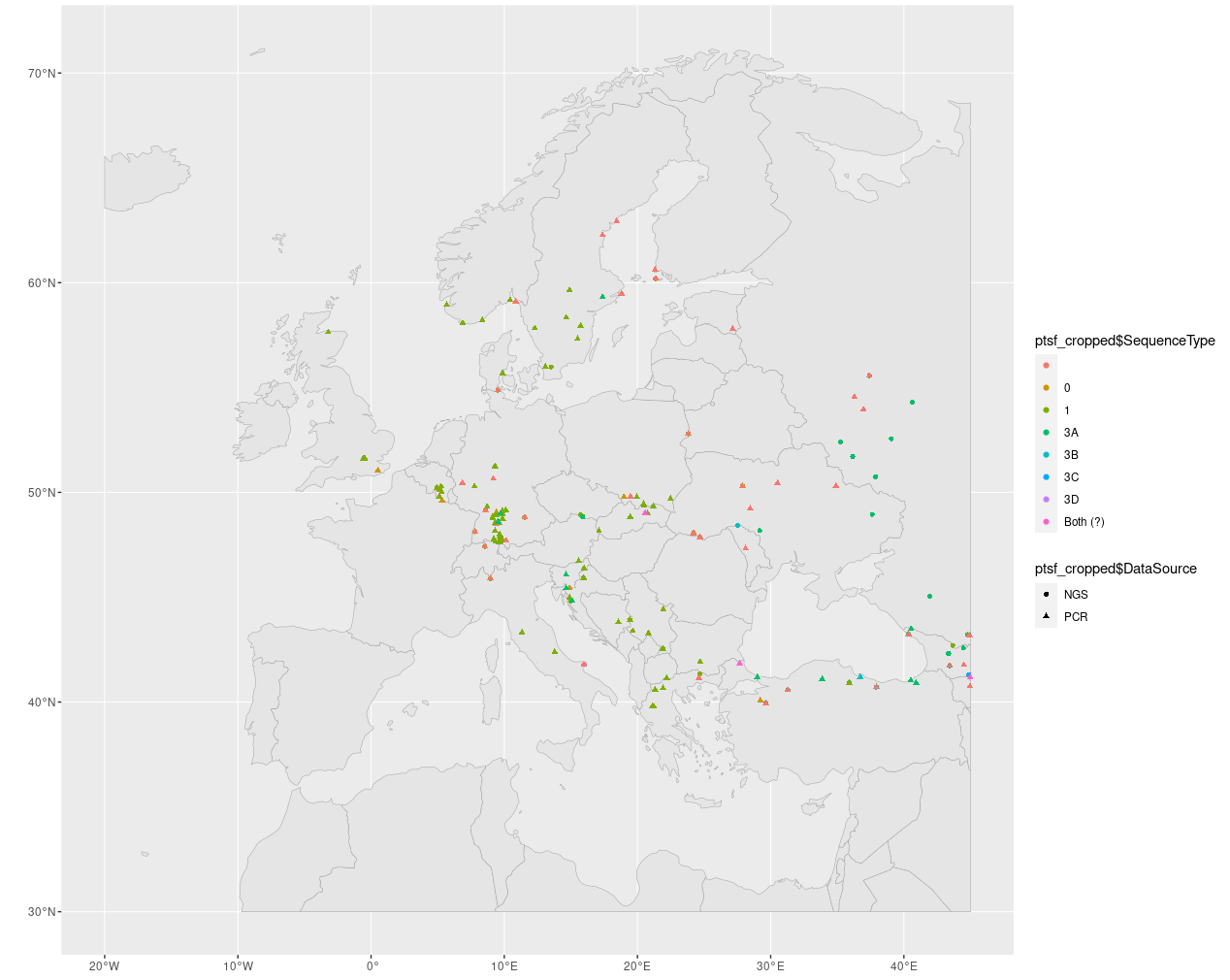
Datenquelle als Form, Sequenztyp als Farbe visualisiert.(Mission accomplished...)
Übungen

