
- ein Ableger von "S"
- eine Statistiksoftware
- eine Programmiersprache
- erweiterbar
- ständig in der Entwicklung
- plattformunabhängig
- gut dokumentiert
- kostenlos und frei erhältlich

- intuitiv bedienbar
- kommerziell unterstützt
Ausdrücke
- Einfache Ausdrücke stellen in R Zahlen oder Zeichenketten (Strings) dar.
- Zeichenketten müssen in einfachen oder doppelten Anführungszeichen stehen.
- Zahlen können für Berechnungen, Zeichenketten für Stringoperationen verwendet werden.
- Berechnungen mit Strings verursachen i. d. R. – wenig überraschend – einen Fehler.
Ausdrücke
- Zahlen:
3 3.14 - Zeichenketten:
"Hallo!" 'Text.' - Rechenausdrücke:
1+1 2*2 1/3 2-1 10^3
Vergleiche
- Vergleiche bestehen aus zwei Ausdrücken oder Objekten und einem Vergleichsoperator.
- Vergleiche liefern als Ergebnis einen Bool'schen Ausdruck: TRUE oder FALSE.
Vergleichsoperatoren sind:
- größer: >
- größer-gleich: >=
- kleiner: <
- kleiner-gleich: <=
- ungleich: !=
- gleich: ==
Vergleiche
- Zahlen:
3>2 2<4 1<=1 2>=1 3==1 - Zeichenketten:
"Hallo!" != 'Text.' "X" == "U" - Bool'sche Ausdrücke:
TRUE == TRUE TRUE != FALSE
Logische Operationen
- Logische Operationen verknüpfen zwei Ausdrücke mit einem logischen Operator.
- Logische Operationen liefern als Ergebnis einen Bool'schen Ausdruck: TRUE oder FALSE.
- Anwendbar auf: Bool'schen Ausdrücke, Zahlen
- Nicht anwendbar auf: Strings
Operatoren sind:
- Und: & oder &&
- Oder: | oder ||
- Nicht: vorangestelltes !
- Exklusiv oder: Funktion xor(a, b)
Logische Operationen
- Und:
TRUE & TRUE TRUE && FALSE - Oder:
TRUE | TRUE TRUE || FALSE - Nicht:
! TRUE - Exklusiv oder:
xor(TRUE, FALSE)
Ausprobieren!
- R gibt es hier: https://www.r-project.org/
- R-Studio gibt es dort: https://www.rstudio.com/
- Beides sollte auf den Kursrechnern bereits vorinstalliert sein.
- Starten und loslegen…
Ausprobieren!
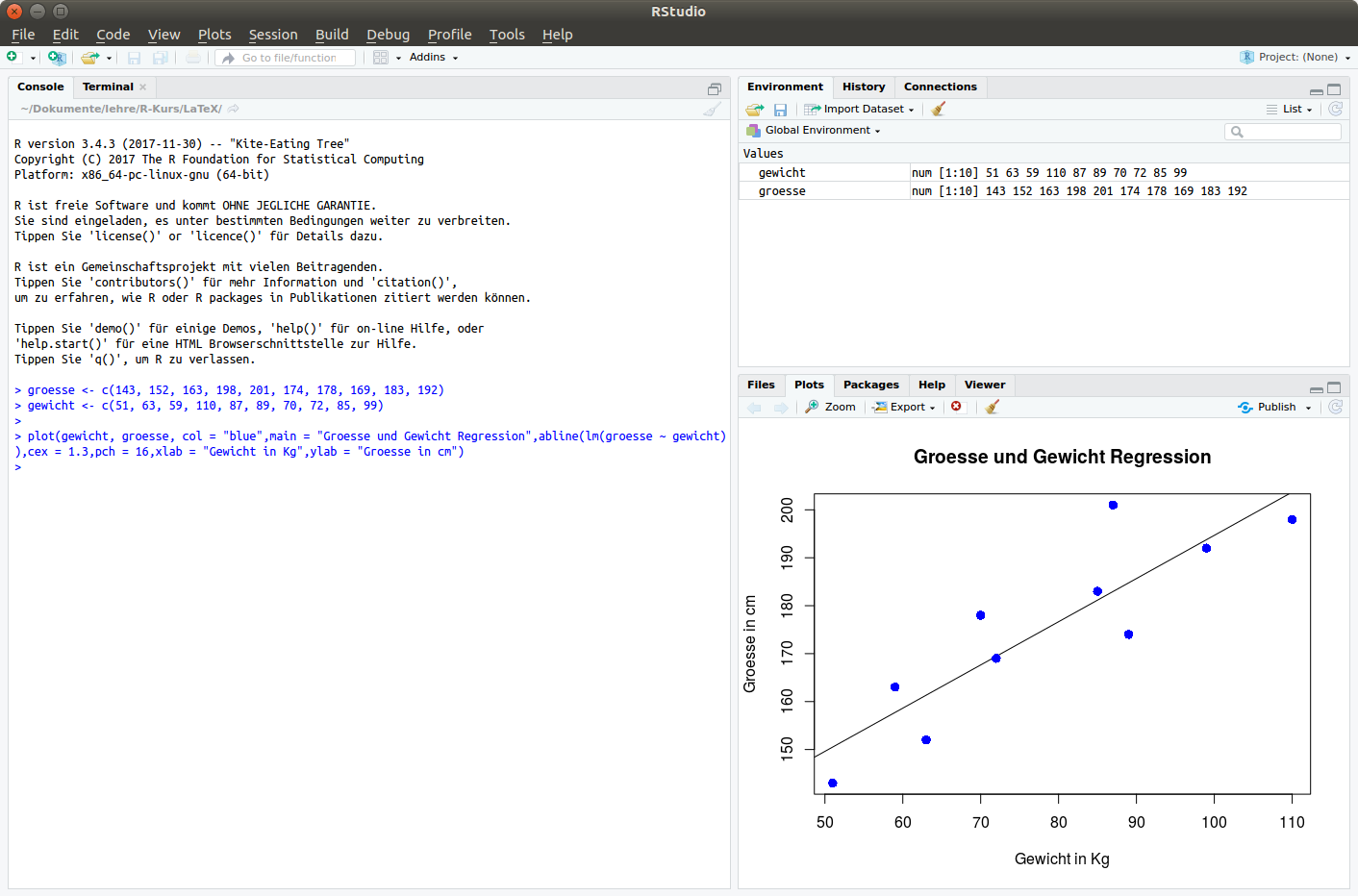
Variablen
- Wie jede Programmiersprache kennt R das Konzept "Variablen".
- Variablen sind mit einem Namen versehene Speichermöglichkeiten für Daten.
- Variablen sind i.d.R. veränderbar.
- Variablentypen sind in R dynamisch, d.h. der Datentyp, den eine Variable aufnehmen kann, muss nicht vorher festgelegt werden.
- Der Zuweisungsoperator ist in R: <- oder = oder ->
Variablen
Zum definieren von Variablen sind Buchstaben, Zahlen, Punkt und Unterstrich im Namen erlaubt, also:
| variable_nummer1. | ok |
| .variable_nummer | ok |
| variable.nummer | ok |
aber
| .1variable_nummer | nicht ok, nach "." darf keine Zahl folgen |
| 1variable_nummer | nicht ok, Name darf nicht mit einer Zahl beginnen |
| _variable_nummer | nicht ok, Name darf nicht mit einem Unterstrich beginnen |
| variable_nummer | nicht ok |
Zuordnung von Variablen
# Zuordnung mit Gleichheitszeichen
> varname.1 = 1
> varname.1
[1] 1
# Zuordnung mit <-
> varname.2 <- 1
> varname.2
[1] 1
# Zuordnung mit ->
> "Kurs" -> varname.3
> varname.3
[1] "Kurs"
Überblick und Aufräumen
Liste aller Variablen
# Die Funktion ls() gibt eine Liste aller benutzten Variablen aus:
> ls()
[1] "alpha" "beta" "df0" "df1" "dg" "g" "x" "m" "M" "pk" "y"
Müll rausbringen…
# Die Funktion rm() loescht ueberfluessige Variabeln
> rm(x)
> ls()
[1] "alpha" "beta" "df0" "df1" "dg" "g" "m" "M" "pk" "y"
# Alles leer raeumen:
> rm(list=ls())
Ausgabe
Die Ausgabe zum Bildschirm oder in eine Datei kann auch mit print() oder cat() erfolgen. Mit cat() können mehrere Ausgaben verbunden werden.
varname1<-'test1'
varname2<-'test2'
print(varname1)
print(varname2)
cat(varname1, varname2)
Hinten und Vorn
head() und tail() ermöglichen die Ausgabe nur des Anfangs oder Endes eines Objekts.
> seq(1,100)
[1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18
[19] 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36
[37] 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54
[55] 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72
[73] 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 89 90
[91] 91 92 93 94 95 96 97 98 99 100
> head(seq(1,100),2)
[1] 1 2
Datentypen
| Beschreibung | Beispiel | Datentyp |
|---|---|---|
| Zeichenfolgen | "R Kurs" | character |
| ganze und reele Zahlen | 1000 | numeric |
| ganze Zahlen | 1L, 2L | integer |
| komplexe Zahlen | 6.023x10exp23 | complex |
| logische Werte | TRUE | logical |
| leere Mengen | NULL | NULL |
Datentypen
> variable1 <- "R Kurs"
# Die Abfrage des Typs erfolgt mit class
> class(variable1)
> cat("Die variable1 hat den Typ", class(variable1), "\n")
Die variable1 hat den Typ character
> variable1 <- 1000L
> cat("Jetzt hat die variable1 den Typ", class(variable1), "\n")
Jetzt hat die variable1 den Typ integer
Type casting
Als Type-casting bezeichnet man die Umwandlung von Datentypen untereinander:
# Mit as.integer kann auch eine variable dem Typ integer zugeordnet werden:
> variable2 <- as.integer(1000)
> class(variable2)
[1] "integer"
Überschreiben
Variablen sind keine Konstanten.
# Variablen koennen ueberschrieben werden
> variable1 <- "Test"
> variable1 <- 10.7
> cat("Die variable1 hat den Typ", class(variable1), ".\n")
Die variable1 hat den Typ numeric.
Typen
Intern werden Daten von R in expliziteren Typen gespeichert:
var1 <- 10.7
var2<-10
var3 <- as.integer(var1)
var4 <- as.integer(var2)
# Im internen Speichermodus entspricht numeric
# double, Abfrage des internen Speichermodus mit typeof
> typeof(var1)
[1] "double"
Welche inneren und äußeren Typen haben die verwendeten Variablen?
Variablen löschen
# Variablen koennen durch rm geloescht werden
> var1 <- 1000
> rm(var1)
> var1
Fehler: Objekt 'var1' nicht gefunden
Funktionen
Viele Funktionen gehören zu R oder zu verschiedenen statistischen Paketen.
Funktionen werden z. B. zum ein- und auslesen von Daten, für statistische Zwecke oder zum Plotten von Daten verwendet.
Benutzerdefinierte Funktionen werden später besprochen.
Funktionen
Mit Funktionen werden bestimmte definierte Aufgaben ausgeführt. In R existieren bereits viele vordefinierte Funktionen, z.B. sum(), mean(), sqrt():
# Summe von Zahlen mit sum()
> sum(1000, 1)
[1] 1001
# Mittelwert von Zahlen eines Vektors mit mean()
# (Vektoren werden spaeter besprochen)
> mean(c(1, 2, 3, 4, 5))
[1] 3
Quadratwurzel
# Bilden der Quadratwurzel
> sqrt(49)
[1] 7
Logarithmen
# Gebraeuchliche Logarithmen
# Logarithmus zur Basis 10
> log10(1000)
[1] 3
# Logarithmus zur Basis 2
> log2(64)
[1] 6
# natuerlicher Logarithmus
> log(100)
[1] 4.60517
# Bilden des Exponenten
> exp(4.60517)
[1] 99.99998
# NB.: Rundungsfehler!!
Hilfe!
R enthält auch eine Gebrauchsanweisung: ?sqrt oder help(sqrt) liefert die Handbuchseite für die Funktion sqrt().
# Was gibt folgendes aus?
help(help)
'q' beendet die Hilfedarstellung.
Datenstrukturen
R organisiert Daten in bestimmten Datenstrukturen,
z.B. Vektoren, Listen, Matrizen
- Vektor
- Matrix
- Array
- Data frame
- List
- Factor
Vektoren
- Vektoren sind elementar in R
- Vektoren bestehen aus einem Element oder mehreren geordneten Elementen
- die Daten in den Vektoren können von verschiedenem Datentyps sein, z.B. numeric, character, logical
- alle Elemente eines Vektors müssen zum selben Datentyp gehören!
Vektoren
- Fast alle Objekte in R sind durch Vektoren dargestellt.
- Vektoren können von beliebigem Datentyp sein, allerdings müssen alle Objekte in einem Vektor von demselben Datentyp sein.
- Die einfachsten Vektortypen in R enthalten nur ein Element.
Vektoren
# So erstellt man einen Vektor des Typs character
> print("Kurs")
[1] "Kurs"
# Der Vektor kann in einer Variablen abgelegt werden
> var1 <- ("Kurs")
[1] "Kurs"
# Abfrage ob var1 ein Vektor ist erfogt durch vorangestelltes "is."
> is.vector(var1)
[1] TRUE
Vektoren
# Ein bestimmter Dateityp kann durch vorangestelltes "as." erzwungen werden
> var2 <- pi
> var3 <- as.integer(var2)
> var3
[1] 3 # Hier erfolgt ein Informationsverlust!
> class(var3)
[1] "integer"
Vektoren
Numerische Vektoren mit mehreren Elementen:
# Einen numerischen Vektor kann man auf unterschiedliche Arten erzeugen
> x <- c(1, 2, 3, 4, 5)
> x
[1] 1 2 3 4 5
> x <-seq(from = 1, to = 5, by = 1))
> x
[1] 1 2 3 4 5
> x <- c(1:5)
> x
[1] 1 2 3 4 5
Vektoren
Die einfachste Art Vektoren in R zu erzeugen ist c (für combine oder concatenate)
# numerischer Vektor
> x <- c(1:100)
# "character" Vector
> x <- c("Ich", "brauche", "eine", "Pause")
> x
[1] "Ich" "brauche" "eine" "Pause"
# Boolean Vektor
> x <- c(FALSE, TRUE, T, F)
> x
[1] FALSE TRUE TRUE FALSE
Vektoren
Gemischte Vektoren:
> x <- c(1, 2, TRUE, "Kaffee")
> x
# Da alle Elemente des Vektors denselben Datentyp besitzen muessen, wird hiermit ein Vektor des Typs "Charakter" erzwungen.
>is.character(x)
[1] TRUE
Vektoren
# Kombinieren von Vektoren
> x <- c(1:5)
> y <- c(x, 6:10)
> y
[1] 1 2 3 4 5 6 7 8 9 10
> length(y)
[1] 10
Vektoren
Für Wiederholung desselben Objektes kann die Funktion rep() verwendet werden
> x <- rep(3, 12)
> x
[1] 3 3 3 3 3 3 3 3 3 3 3 3
> length(y)
[1] 12
Rechnen mit Vektoren
Das Rechnen mit Vektoren erfolgt elementweise:
> x <- c(1:5)
> y <- x*2
> y
[1] 2 4 6 8 10
> z <- x + x
> z
[1] 2 4 6 8 10
> z <- x ^2
> z
[1] 1 4 9 16 25
> mean(x)
[1] 3
> sum(x)
[1] 15
Indizieren von Vektoren
Syntax: Objekt[Index-Vektor]
> x <- c(1:5)
> x[3] # indiziert das 3. Element des Vektors
[1] 3
> x[c(4, 1)] # indiziert das 4. und 1. Element des Vektors, Reihenfolge beachten!
[1] 4 1
> x[-c(4, 1)] # schliesst das 4. und 1. Element des Vektors aus
[1] 2 3 5
Andere Datenstrukturen
Alle Datenstrukturen in R gehören zu bestimmten Klassen. Vektoren sind die wichtigste Datenstruktur in R. Alle anderen Objekte können als Kombination von Vektoren in eine bestimmten Struktur verstanden werden.Matrizen
Die Daten in Matrizen sind in zweidimensionaler Form organisiert. Alle Elemente einer Matrix müssen denselben Datentyp enthalten. Meist werden Elemente des Typs numeric in Matrizen verwendet.Datensätze - data frames
Dataframes sind ähnlich den Matrizen, jedoch können verschiedene Spalten unterschiedlichen Datentypen angehören.Listen
Listen sind R Objekte, die Elemente verschiedener Typen enthalten, z.B. Zahlen, Vektoren, oder auch andere Listen.Faktoren
Faktoren sind R Objekte, die Daten kategorisieren und in Leveln behandeln, z.B. weiblich - männlich, oder Wildtyp - Mutante.Arrays
Arrays sind Objekte, um Daten in mehr als zwei Dimensionen zu speichern. Ein Array der Dimension (2, 3, 4) stellt sich dar, als 4 Matrizen mit je zwei Zeilen und 3 Spalten. Arrays werden wir in diesem Kurs nicht weiter behandeln.Datentypen
| Typ | Dim | Mix | Named |
|---|---|---|---|
| Vector | 1 | FALSE | FALSE |
| Matrix | 2 | FALSE | TRUE |
| Data frame | 2 | TRUE (spaltenweise) | TRUE |
| List | 1 (but...) | TRUE | FALSE |
| Factor | 1 | FALSE | FALSE |
| Array | n | FALSE | TRUE |
Erstellen einer Matrize
Matrizen können mit der Funktion matrix() erstellt werden. Syntax: matrix(data, nrow, ncol, byrow, dimnames)- data Daten als Input-Vektor, die die Elemente der Matrize bilden werden
- nrow Anzahl Zeilen
- ncol Anzahl Spalten
- byrow Wenn byrow TRUE ist, werden die Elemente des Vektors zeilenweise eingetragen
- dimnames bezeichnet die Namen der Zeilen und Spalten
Erstellen einer Matrize
# Elemente werden Spaltenweise eingetragen
> x <- matrix(1:10, nrow = 2, ncol = 5)
> x
[,1] [,2] [,3] [,4] [,5]
[1,] 1 3 5 7 9
[2,] 2 4 6 8 10
# Elemente werden Zeilenweise eingetragen
> x <- matrix(1:10, nrow = 2, ncol = 5, byrow = TRUE)
> x
[,1] [,2] [,3] [,4] [,5]
[1,] 1 2 3 4 5
[2,] 6 7 8 9 10
Erstellen einer Matrize
# Jetzt werden den Zeilen und Spalten Namen gegeben
> rownames <- c("Zeile1", "Zeile2")
> colnames <- c("Spalte1", "Spalte2", "Spalte3", "Spalte4", "Spalte5")
> y <- matrix(1:10, nrow = 2, ncol = 5,
byrow = TRUE, dimnames = list(rownames, colnames))
> y
Spalte1 Spalte2 Spalte3 Spalte4 Spalte5
Zeile1 1 2 3 4 5
Zeile2 6 7 8 9 10
Indizieren von Matrizen
Elemente einer Matrix können adressiert werden, indem man den Spalten- und Zeilenindex der Elemente angibt.
# Auslesen des Elements in Spalte3, Zeile 2
> print(y[2,3])
> [1] 8
# Auslesen der Elemente von Spalte4
> print(y[,4])
Zeile1 Zeile2
4 9
Rechnen mit Matrizen
Dies kann natürlich auch für Berechnungen genutzt werden
# Hier wird der Mittelwert aller Elemente in Zeile1 in Spalte1 bis Spalte3 berechnet
> print(mean(y[1,c(1, 2, 3)]))
Dataframes
Dataframes sind den Matrizen ähnlich, jedoch können unterschiedliche Datentypen verwendet werden. In Matrizen ist dies nicht erlaubt. Daher werden Zahlen im folgenden Beispiel als Datentyp "character" behandelt.Dataframe
# In einer Matrize haben alle Elemente denselben Datentyp
> x <- matrix(c(1:5, c("a", "b", "c", "d", "e")), 5, 2)
> x
[,1] [,2]
[1,] "1" "a"
[2,] "2" "b"
[3,] "3" "c"
[4,] "4" "d"
[5,] "5" "e"
> y <- x[2,1]
> class(y)
[1] "character"
Gemischte Dataframes
In einem dataframe können Elemente unterschiedlichen Datentyps kombiniert werden. Die Spaltennamen (colnames) sollen nicht leer sein, die Zeilennamen (rownames) sollen eindeutig sein.
# Wir definieren dieselben Daten als Dataframe
> d <- data.frame(ersteSpalte = 1:5, zweiteSpalte = c("a", "b", "c", "d", "e"))
ersteSpalte zweiteSpalte
1 1 a
2 2 b
3 3 c
4 4 d
5 5 e
Dataframe Beispiel
Im Folgenden ein Beispiel zur Samenentwicklung in 5 Pflanzen einer Mutanten Arabidopsis Linie. Angegeben werden sollen Pflanzen Nummer, Anzahl gut entwickelter Samen, Anzahl aller Samen.
# Wir definieren das Dataframe
> data <- data.frame(Pflanzen_Nr =
c("At1", "At2", "At3", "At4", "At5"),
fertile_Samen = c(122, 345, 215, 90, 77),
StringsAsFactors = FALSE)
Pflanzen_Nr fertile_Samen
1 At1 122
2 At2 345
3 At3 215
4 At4 90
5 At5 77
Dataframe Struktur
Mit der str() Funktion kann die Struktur des Dataframes angesehen werden
> str(dfrm)
'data.frame': 5 obs. of 2 variables:
$ Pflanzen_Nr : chr "At1" "At2" "At3" "At4" ...
$ fertile_Samen: num 122 345 215 90 77
str() funktioniert übrigens nicht nur mit Dataframes.
Dataframe Zusammenfassung
Die Funktion summary() gibt die statistische Zusammenfassung der Daten wieder
> summary(data)
Pflanzen_Nr fertile_Samen
Length:5 Min. : 77.0
Class :character 1st Qu.: 90.0
Mode :character Median :122.0
Mean :169.8
3rd Qu.:215.0
Max. :345.0
Dataframe erweitern (Spalten)
Einem Dataframe können auch Daten hinzugefügt werden. In diesem Fall sollen die Blühzeiten in Tagen ergänzt werden. Hier wird eine neue Spalte hinzugefügt.
> data$Bluehzeit_Tage <- c(5, 7, 6, 10, 8)
> data <- data
> data
Pflanzen_Nr fertile_Samen Bluehzeit_Tage
1 At1 122 5
2 At2 345 7
3 At3 215 6
4 At4 90 10
5 At5 77 8
Dataframe erweitern (Zeilen)
Es können auch neue Zeilen hinzugefügt werden, in diesem Beispiel die Daten der Pflanzen At6 bis At8. Hierbei wird der Befehl rbind() benutzt. Die neuen Zeilen müssen im selben Format sein, wie das ursprüngliche Dataframe.Dataframe erweitern (Zeilen)
# Zunaechst definieren wir ein neues Dataframe in demselben Format wie data
newdata <- data.frame(Pflanzen_Nr = c("At6", "At7", "At8"),
fertile_Samen = c(99, 45, 102), Bluehzeit_Tage = c (7, 11, 13),
stringsAsFactors = FALSE)
# Jetzt werden beide dataframes verbunden
> data_final <- rbind(data, newdata)
> print(data_final)
Pflanzen_Nr fertile_Samen Bluehzeit_Tage
1 At1 122 5
2 At2 345 7
3 At3 215 6
4 At4 90 10
5 At5 77 8
6 At6 99 7
7 At7 45 11
8 At8 102 13
Indizieren von Dataframes
Das Indizieren von Dataframes erfolgt ähnlich dem Indizieren von Matrizen.
# Hier werden die ersten beiden Zeilen des dataframes data_final extrahiert
> print(data_final[c(1, 2),])
Pflanzen_Nr fertile_Samen Bluehzeit_Tage
1 At1 122 5
2 At2 345 7
# Hier adressieren wir die Bluehzeit_Tage der Pflanzen At5 und At8
> print(data_final[c(5, 8), c(1, 3)])
Pflanzen_Nr Bluehzeit_Tage
5 At5 8
8 At8 13
Listen
Listen werden mit der list() Funktion erstellt. Sie können als Sammelobjekt für verschiedene Komponenten angesehen werden, die aus unterschiedlichen Datentypen bestehen können.
# Wir definieren eine Liste, die einen Vektor und eine Matrix enthaelt.
# Beispiel ist die Expression von 3 Genen in drei Replikaten.
> neueListe <- list(c("WUSCHEL", "CLAVATA3", "TOPLESS"),
expr = matrix(c(101, 59, 120, 289, 310, 278, 789, 1020, 812),
nrow = 3, ncol = 3, byrow = TRUE))
> neueListe
[[1]]
[1] "WUSCHEL" "CLAVATA3" "TOPLESS"
$expr
[,1] [,2] [,3]
[1,] 101 59 120
[2,] 289 310 278
[3,] 789 1020 812
Listen können auch selbst wiederum Listen enthalten.
Faktoren
Mit Faktoren werden Daten bestimmten Kategorien oder Leveln zugeordnet, was bei der statistischen Datenauswertung eine große Rolle spielt. Zum Beispiel kann man die Faktoren-Level "weiblich - männlich", "Wiltyp - Mutante" oder auch "kein Stress - Stress" definieren. Faktoren werden mit dem Befehl factor() erzeugt.Faktoren
# Wir erzeugen einen Vektor mit den Kategorien
> genotype <- c("wildtype", "wildtype", "mutant", "wildtype", "mutant")
# Daraus wird ein Factor mit zwei Leveln:
> factor(genotype)
[1] wildtype wildtype mutant wildtype mutant
Levels: mutant wildtype
Flusskontrolle und Schleifen
Kontrollkonstrukte und Schleifen sind wichtig in allen Programmiersprachen. Sie steuern den Programmablauf indem sie Fallunterscheidungen und Wiederholungen ermöglichen. Die wichtigsten Konstrukte für bedingte Anweisungen in R sind:
# Ist die Bedingung wahr (TRUE) wird Ausdruck1 ausgewertet,
# wenn die Bedingung falsch (FALSE) ist Ausdruck 2. Der andere Ausdruck wird jeweils
# nicht beruecksichtigt.
(1) if(Bedingung){Ausdruck1} else{Ausdruck2}
(2) ifelse(Bedingung, Ausdruck1, Ausdruck2)
Bedingte Ausführung
Ein Beispiel:
> Uhrzeit <- "9 Uhr"
> Fruehstueck <- "9 Uhr"
> Mittagspause <- "12 Uhr"
> if(Uhrzeit == Mittagspause || Uhrzeit == Fruehstueck) {print("Pause")}
else{print("Weiter arbeiten")}
[1] "Weiter arbeiten"
Ein Beispiel für ifelse
ifelse() ist eine Funktion, die eine Bedingung und zwei alternative Ausdrücke annimmt. Sie ist vektorisiert, kann also auf einen Vektor angewendet werden.
> x <- c(1:10)
> ifelse(x > 5, ">5", "<=5")
[1] "<=5" "<=5" "<=5" "<=5" "<=5" ">5" ">5" ">5" ">5" ">5"
Schleifen
Schleifen führen bestimmte Funktionen wiederholt aus. Es gibt verschiedene Möglichkeiten Schleifen in R zu definieren:| repeat{Ausdruck} | Wiederholung des Ausdrucks, erfolgt bis die Schleife angehalten wird |
| while(Bedingung){Ausdruck} | Wiederholung des Ausdrucks solange die Bedingung erfüllt ist |
| for(i in M){Ausdruck} | Wiederholung des Ausdrucks für jedes i der Menge M |
| next | Sprung in den nächsten Wiederholungsschritt |
| break | sofortiger Abbruch der Schleife |
repeat Beispiel
# In diesem Beispiel wird 1 zu i addiert, bis der Wert 10 erreicht ist
> i <- 0
> repeat{
+ i <- i+1
+ print(i)
+ if(i == 10) break
+ }
[1] 1
[1] 2
[1] 3
[1] 4
[1] 5
[1] 6
[1] 7
[1] 8
[1] 9
[1] 10
while Beispiel
# Beispiel zur Benutzung von while
# (bei einzeiligen Ausdruecken koennen die geschweiften Klammern unterbleiben.)
> i <- 0
> while(i < 10)
+ i <- i +1
> i
[1] 10
for Beispiel
> i <- 0
> x <- c(1:10)
> for(i in x)
+ print(i + 1)
[1] 2
[1] 3
[1] 4
[1] 5
[1] 6
[1] 7
[1] 8
[1] 9
[1] 10
[1] 11
Ein- und Ausgabe von Dat(ei)en
R kann Daten von ausserhalb der R Umgebung einlesen und ebenso kann eine Datenausgabe erfolgen.
Ein-/Ausgabe von Dateien
Wichtig dafür ist, zu definieren und zu wissen in welchem Ordner gearbeitet wird. Zum setzen des Arbeitsordners wird der Befehl setwd() (für set working directory) benötigt. Will man nachfragen, in welchem Arbeitsordner man sich befindet, benötigt man getwd().
Die einfachste Funktion Daten in R einzulesen ist read.table(). Die Funktion ist geeignet zum Einlesen eines Datensatzes (dataframe), der in Tabellenform gespeichert ist.
Ein-/Ausgabe von Dateien
Wichtige Argumente dieser Funktion sind:- file – Dateiname und Pfad (letzerer nur, wenn nicht im working directory vorhanden)
- header – deklariert, ob Spaltennamen vorhanden sind, die default Einstellung ist FALSE
- sep – definiert die Trennzeichen zwischen den Spalten. Default ist ein Leerzeichen oder Tab (" ", "\t")
- dec – definiert, welches Trennzeichen Dezimalstellen trennt, die Voreinstellung ist "."
- Informationen finden sich in der Hilfe ?read.table
Einlesen von Daten
> ?read.table()
read.table(file, header = FALSE, sep = "", quote = "\"'",
dec = ".", numerals = c("allow.loss", "warn.loss", "no.loss"),
row.names, col.names, as.is = !stringsAsFactors,
na.strings = "NA", colClasses = NA, nrows = -1,
skip = 0, check.names = TRUE, fill = !blank.lines.skip,
strip.white = FALSE, blank.lines.skip = TRUE,
comment.char = "#",
allowEscapes = FALSE, flush = FALSE,
stringsAsFactors = default.stringsAsFactors(),
fileEncoding = "", encoding = "unknown", text, skipNul = FALSE)
Schreiben von Dateien
Zur Dateiausgabe wird die Funktion write.table() verwendet.
write.table(x, file = "", append = FALSE, quote = TRUE, sep = " ",
eol = "\n", na = "NA", dec = ".", row.names = TRUE,
col.names = TRUE, qmethod = c("escape", "double"),
fileEncoding = "")
?write.table
Austausch mit MS Excel
Für den Datenaustausch mit Microsoft Excel bieten sich CSV-Dateien an. Die relevanten R-Funktionen lauten:
read.csv(file, header = TRUE, sep = ",", quote = "\"",
dec = ".", fill = TRUE, comment.char = "", ...)
read.csv2(file, header = TRUE, sep = ";", quote = "\"",
dec = ",", fill = TRUE, comment.char = "", ...)
read.delim(file, header = TRUE, sep = "\t", quote = "\"",
dec = ".", fill = TRUE, comment.char = "", ...)
read.delim2(file, header = TRUE, sep = "\t", quote = "\"",
dec = ",", fill = TRUE, comment.char = "", ...)
write.csv(...)
write.csv2(...)
Übungsdaten in R
Als Beispiel rufen wir den berühmten Datensatz "iris" auf.
?iris
iris {datasets}R Documentation
Edgar Anderson's Iris Data
Description
This famous (Fisher's or Anderson's) iris data set gives the measurements in
...
> head(iris)
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
Sepal.L.. Sepal.W.. Petal.L.. Petal.W.. Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
> write.table(iris, file = "iris_data.txt", sep = " ")
Iris
Denselben Datensatz können wir mit read.table() wieder einlesen:
> test <- read.table("iris_data.txt", header = TRUE, sep = " ")
> head(test)
Sepal.L.. Sepal.W.. Petal.L.. Petal.W.. Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa